|
[GT] 고도로 정밀해진 휴대용 DNA 시퀀서 | |||
| 덴마크 올보르 대학교(Aalborg University)의 연구원들은 최... |


  |
 |  |
 |
[GT] 고도로 정밀해진 휴대용 DNA 시퀀서
게놈 서열은 유기체의 정체성, 조상, 강점 및 취약성을 포함하여 유기체에 대한 많은 것을 드러낼 수 있다. 과학자들은 이 정보를 사용하여 특정 환경에 살고 있는 미생물을 더 잘 이해하고 진단 도구와 치료법을 개발한다. 그러나 정확한 휴대용 DNA 시퀀서가 없으면 현장이나 소규모 실험실에서 연구를 수행할 때 중요한 유전적 세부 사항을 놓칠 수 있다.
덴마크 올보르 대학교(Aalborg University)의 연구원들은 최근 널리 사용되는 휴대용 DNA 시퀀서에서 발생하는 거의 모든 시퀀싱 오류를 제거하는 간단한 방법을 시연했다. 이는 잠재적으로 실험실 밖에서 일하는 과학자들이 미생물을 보다 효율적으로 연구하고 추적할 수 있게 해준다.
특수 분자 태그를 사용하여 연구팀은 한 번에 많은 긴 DNA를 시퀀싱하는 경우에도 옥스포드 나노포어 테크놀로지(Oxford Nanopore Technologies)에서 만든 미니온(MinION) 장치의 일반적인 5~15% 오류율을 0.005% 미만으로 줄일 수 있었다.
미니온은 대규모 실험실의 한계에서 DNA 시퀀싱을 해방함으로써 이미 유전체학 분야에 혁명을 일으킨 장치이다. 그러나 지금까지 연구자들은 기본 오류율이 상당히 높기 때문에 많은 상황에서 이 장치에 의존할 수 없었다.
이에 올보로 대학 연구팀은 미니온과 같은 긴 판독 DNA 시퀀싱 플랫폼을 1000배 이상 더 정확하게 만들 수 있는 바코드 시스템을 만들었다. 이 바코드로 표적 분자에 태그를 지정한 후, 연구자들은 표준 PCR 기술을 사용하여 태그가 지정된 분자를 증폭하거나 여러 복사본을 만들고 생성된 DNA를 시퀀싱하는 등 평소와 같이 진행한다.
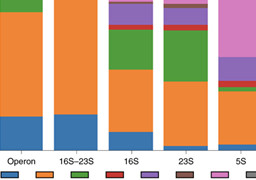
이후 연구자들은 바코드를 사용하여 시퀀싱 데이터에서 관련 DNA 단편을 쉽게 식별하고 그룹화하여 궁극적으로 기존 기술이 처리할 수 있는 것보다 최대 10배 더 긴 단편에서 거의 완벽한 시퀀스를 생성할 수 있었다. 더 긴 DNA 스트레치는 약간의 유전적 변이를 감지하고 게놈을 고해상도로 조립할 수도 있다.
이 방법의 우수한 점은 증폭될 수 있는 모든 관심 유전자에 적용할 수 있다는 것이다. 이는 암 연구, 식물 연구, 인간 유전학, 마이크로바이옴 과학과 같이 고도의 정확도와 긴 게놈 정보의 결합이 중요한 분야에서 매우 유용할 수 있음을 의미한다.
연구자들은 현재 메트로 밴쿠버와 협력하여 물과 폐수에서 미생물을 거의 실시간으로 탐지할 수 있는 확장 버전을 개발하고 있다.
Reference:
NATURE METHODS, January 11, 2021, “High-Accuracy Long-Read Amplicon Sequences Using Unique Molecular Identifiers with Nanopore or PacBio Sequencing,” by Søren M. Karst, et al. © Springer Nature Limited. All rights reserved.
To view or purchase this article, please visit:
High-accuracy long-read amplicon sequences using unique molecular identifiers with Nanopore or PacBio sequencing chr(124)_pipe Nature Methods
 |  |
 |
[Original Text]
Genome sequences can reveal a great deal about an organism, including its identity, its ancestry, its strengths and its vulnerabilities. Scientists use this information to better understand the microbes living in a particular environment, as well as to develop diagnostic tools and treatments. But without accurate portable DNA sequencers, crucial genetic details could be missed when research is conducted out in the field or in smaller laboratories.
Researchers at Aalborg University recently demonstrated a simple way to eliminate almost all sequencing errors produced by a widely used portable DNA sequencer, potentially enabling scientists working outside the lab to study and track microorganisms more efficiently.
Using special molecular tags, the team was able to reduce the normal five-to-15 percent error rate of the MinION device made by Oxford Nanopore Technologies to less than 0.005 percent, even when sequencing many long stretches of DNA at a time.
The MinION has already revolutionized the field of genomics by freeing DNA sequencing from the confines of large laboratories. But until now, researchers haven’t been able to rely on the device in many situations because of its fairly high out-of-the-box error rate.
So the Aalborg University team created a unique barcoding system that can make long-read DNA sequencing platforms like the MinION over 1000 times more accurate. After tagging the target molecules with these barcodes, researchers proceed as they usually would - amplifying, or making multiple copies of, the tagged molecules using the standard PCR technique and sequencing the resulting DNA.
The researchers can then use the barcodes to easily identify and group relevant DNA fragments in the sequencing data, ultimately producing near-perfect sequences from fragments that are up to 10 times longer than conventional technologies can process. Longer stretches of DNA allow the detection of even slight genetic variations and the assembly of genomes in high resolution.
A beautiful thing about this method is that it is applicable to any gene of interest that can be amplified. This means that it can be very useful in fields such as cancer research, plant research, human genetics, and microbiome science where the combination of high-accuracy and long- range genomic information is valuable.
The researchers are currently collaborating with Metro Vancouver to develop an expanded version of the method that permits the near-real-time detection of microorganisms in water and wastewater.
Reference:
NATURE METHODS, January 11, 2021, “High-Accuracy Long-Read Amplicon Sequences Using Unique Molecular Identifiers with Nanopore or PacBio Sequencing,” by Søren M. Karst, et al. © Springer Nature Limited. All rights reserved.
To view or purchase this article, please visit:
High-accuracy long-read amplicon sequences using unique molecular identifiers with Nanopore or PacBio sequencing chr(124)_pipe Nature Methods
_아침독서상단(2).jpg)
.jpg)
.jpg)
.jpg)